RNA
Ribo Nükleik asit veya RNA bir nükleik asittir, nükleotitlerden oluşan bir polimerdir. Her nükleotit bir azotlu baz, bir riboz şeker ve bir fosfattan oluşur. RNA pek çok önemli biyolojik rol oynar, bunların arasında DNA'da taşınan genetik bilginin proteine çevirisi (translasyon) ile ilişkili çeşitli süreçlerde de yer alır. RNA tiplerinden olan mesajcı RNA, DNA'daki bilgiyi protein sentez yeri olan ribozomlara taşır, ribozomal RNA ribozomun en önemli kısımlarını oluşturur, taşıyıcı RNA ise protein sentezinde kullanılmak üzere kullanılacak aminoasitlerin taşınmasında gereklidir. Ayrıca çeşitli RNA tipleri genlerin ne derece aktif olduğunu düzenlemeye yarar.
RNA, DNA'ya çok benzer olmakla beraber bazı yapısal ayrıntılarında farklılık gösterir. Hücre içinde RNA genelde tek zincirli, DNA ise genelde çift zincirlidir. RNAnükleotitleri riboz içerirler, DNA ise deoksiriboz (bir oksijeni atomu eksik olan bir riboz türü) vardır. DNA'da bulunan timin bazı yerine RNA'da urasil vardır ve genelde RNA'daki bazlar ayrıca kimyasal modifikasyona uğrar. RNA, RNA polimeraz enziminin DNA'yı okuması (transkripsiyonu) ile sentezlenir ve ardından başka enzimler tarafından işlenerek değişime uğrar. Bu RNA işleyici enzimlerin bazıları kendi RNA'larını içerirler.
Yapısı
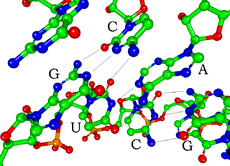
RNA'daki her nükleotit bir riboz şekeri içerir, bunun karbonları 1' ila 5' olarak numaralandırılır. 1' konumuna bir baz bağlıdır, genelde adenin (A), sitozin (C), guanin (G) veya urasil (U). İki riboz arasında bir fosfat grubu vardır, bu fosfat bir ribozun 3' konumuna, öbür ribozun ise 5' konumuna bağlıdır. Fizyolojik pH'de fosfat grubu negatif bir yük taşıdığı için RNA yüklü bir moleküldür (polianyon). Bazı bazlar arasında hidrojen bağları oluşabilir: sitozin ve guanin, adenin ve urasil ve bazen guanin ve urasil arasında bu tür bağlar oluşur.[1] Ancak, RNA zinciri çeşitli şekiller alabildiği için bunlardan başka baz-baz etkileşimleri de mümkündür, örneğin bir grup adenin birbiriyle bağlanarak RNA zincirinde bir tümsek oluşturabilir,[2] veya GNRA dörtlüsü'nde bir guanin-adenin etkileşimi olur.[1]
RNA'yı DNA'dan farklı kılan önemli bir fark, riboz şekerin 2' konumundaki hidroksil grubudur. Bu fonksiyonel grubun varlığı c3'-endo şeker konformasyonunu zorunlu kılar, buna karşın DNA'nın deoksiriboz şekerinin C2'-endo konformasyonu vardır. Bunun sonucu olarak RNA'nin çifte sarmallı kısımları A-şekilli olur, DNA'da yaygın olarak görülen B şekilli sarmaldan farklı olarak.[3] A-şekilli sarmalın büyük oyuğu B şekilli sarmala kıyasla daha derin ve dardır, küçük oyuğu ise sığ ve geniştir.[4] 2' hidroksil grubunun ikinci bir etkisi ise, RNA'nın esnek olan bölgelerinde (yani çift sarmal oluşturmamış kısımlarında) bu hidroksil grubunun yanındaki fosfodiester bağa saldırıp şeker-fosfat zincirin kesilmesine neden olabilmesidir.[5]
RNA transkripsiyonu sırasında sadece dört baz kullanılır (adenin, sitozin, guanin ve urasil)[6] ama ergin RNA'larda pek çok değişime uğramış şeker ve baz vardır. Psödouridin (Ψ) adlı nükleozitte urasil ile riboz arasındaki bağ, bir C-N bağından C-C bağına değişmiştir. Psödouridin ve ribotimidin (T) beraberce çeşitli RNA'larda görülür, özellikle tRNA'ların TΨC ilmiğinde.[7] Değişime uğramış bazlardan bir diğeri olan hipoksantin, deamine olmuş bir guanin bazıdır, nükleozit hali inosin olarak adlandırılır. Genetik kodun değişkenliğinin açıklanmasında inosin anahtar bir rol oynar.[8] Değişime uğramış 100'e aykın nükleozit bilinmektedir,[9] bunların arasında psödouridin ve 2'-O-metilribozlu nükleozitler en yaygın olanlarıdır.[10] Bu modifikasyonların çoğunun işlevi bilinmemektedir. Ancak ribozomal RNA'da çoğu transkripsiyon sonrası modifikasyon, ribozomun en işlevsel bölgelerinde, örneğin peptidil transferaz merkezinde ve altbirim arayüzlerinde yer alması kayda değerdir, bu nedenle bu modifikasyonların normal fonksiyon için gerekli olduğu anlaşılmaktadır.[11]
Tek iplikli bir RNA'nın işlevsel şekli, tıpkı proteinlerde olduğu gibi, çoğu zaman belli bir üçüncül yapı gerektirir. Bu yapının iskeleti, molekülün içindeki bazlar arasındaki hidrojen bağlarıyla ortaya çıkar. Bu şekilde firkete yapısı, tümsek ve ilmik gibi belli ikincil yapı elemanlarından oluşan bölgeler ortaya çıkar.[12] Bir RNA dizisinin nasıl bir üç boyutlu şekil alacağının tahmini halen aktif bir araştırma konusudur.
DNA ile kıyaslama

RNA ve DNA, üç ana özellikleriyle birbirlerinden farklılık gösterirler. Birincisi, DNA çift iplikli olmasına karşın, coğu biyolojik fonksiyonunda RNA tek ipliklidir, ve DNA'dan çok daha kısadır. İkincisi, DNA'yı oluşturan şeker molekülleri deoksiriboz, RNA'yı oluşturanlar ise ribozdur, yani DNA'da pentoz halkasının 2' konumunda bir hidroksil grubu yoktur, RNA'da ise pentoz halkasının iki hidroksil grubu vardır. Rna'da fazladan bulunan hidroksil grupları, hidroliz nedeniyle onun DNA'dan daha az dayanıklı olmasına neden olur. Üçüncüsü, adenin bazını tümleyen baz DNA'daki gibi timin değil, urasildir.
RNA genelde tek iplikli olmasına rağmen, çoğu RNA molekülü katlanarak baz eşleşmesi ile çift sarmallı bölgeler oluşturur. DNA'dan farklı olarak RNA'lar uzun çift iplikli sarmallar değil, birbirine sıkıca sokulmuş kısa sarmallardan oluşur. Bu baz eşleşmeleri RNA molekülüne belli bir şekil verir ve bazların fonksiyonel grupların bir araya gelmesi sonucu reaktif özelliğe sahip olan yapılar ortaya çıkar. Bu sayede RNA, bir enzim gibi, kimyasal katalizör olarak işlev verebilir.[13] Örneğin, peptit bağını oluşturan bir enzim olan ribozomun aktif merkezi tamamen RNA'dan oluşmaktadır.[14]
Sentez
RNA sentezi genelde DNA'yı bir şablon olarak kullanarak, RNA polimeraz enzimi tarafından katalizlenir. Sentezin başlaması DNA üzerinde, RNA'ya yazılacak bölgenin hemen "yukarı" tarafındaki bir diziye enzimin bağlanması ile olur. DNA çifte sarmalı, RNA polimerazın helikaz aktitivitesi ile açılır. Sonra, enzim DNA'nın şablon ipliği üzerinde 3'- 5' doğrultusunda ilerler ve bunun dizisini tümleyici bir diziye sahip bir RNA zincirini 5'-3' doğrultusunda sentezler. DNA üzerinde bulunan belli bir dizi, RNA sentezinin nerede sona ereceğini belirler.[15]
Yukarıda anlatılan DNA'ya bağımlı RNA polimeraz'dan farklı olarak bir de RNA'ya bağımlı RNA polimerazlar vardır, bunlar yeni bir RNA zincirini sentezlemek için şablon olarak bir RNA zinciri kullanırlar. Örneğin, bir grup RNA virüsleri (çiçek virüsü gibi) bu enzimi kullanarak genetik malzemelerini çoğaltırlar.[16] Ayrıca, RNA'ya bağımlı RNA polimeraz çoğu canlıda RNA enterferans yolunda görev alır.[17]
RNA Tipleri
Genel bakış
Mesajci RNA (mRNA) DNA'daki bilgiyi protein sentezi (translasyon) için ribozomlara taşıyan RNA'dır. mRNA'daki kodlayıcı nükleotit dizisi ondan üretilen proteinin amino asit dizisini belirler.
RNA genleri proteine çevrilmeyen, RNA kodlayan genlerdir, bunlar kodlamayan RNA veya küçük RNA olarak adlandırılır. Kodlamayan RNA'lar intronlardan da ortaya çıkabilir.[18] Kodlamayan RNA'ların en belirgin örnekleri taşıyıcı RNA (tRNA) ve ribozomal RNA (rRNA)'dır, bunların ikisi de translasyon sürecinde rol oynarlar.[19] Gen düzenlemesi, RNA işlenmesi ve başka işleveleri olan RNA'lar da vardır. Bazı RNA'lar, başka RNA'ların kesilmesi ve birleştirilmesi (ligasyon)[20] ve ribozomda peptit bağı oluşumu[14] gibi kimyasal tepkimeleri katalizleme yeteneğine sahiptir; bu tip RNA'lar ribozim olarak adlandırılırlar.
Çift iplikli RNA (İng. double stranded RNA 'nın kısaltması olan dsRNA olarak değinilir), birbirini tümleyici iki iplikten oluşmuş RNA'dır, bu bakımdan şekli DNA'ya benzer. Çift iplikli RNA, bazı virüslerin (çift iplikli virüslerin) genetik malzemesini oluşturur. Ökaryotlarda, virüs RNA'sına benzeyen uzun çift iplikli RNA'lar RNA enterferansını harekete geçirir. RNA enterferansında, siRNA (İng. small interfering RNA, kısa enterfreanscı RNA) olarak adlandırılan kısa çift iplikli RNA'lar gen ifadesini susturur.[21]
Translasyonda
Mesajcı RNA (mRNA) bir proteinin amino asit dizisi hakkında bilgiyi protein sentez yeri olan ribozomlara taşır. Bu bilgi, her üç nükleotit (bir kodon) bir amino asite karşılık gelecek şekilde şifrelenmiştir. Ökaryotlarda bir öncül (prekürsör) mRNA (pre-mRNA) DNA'dan yazıldıktan sonra ergin mRNA'ya dönüştürülür. Bu işlem sırasında pre-mRNA'nın protein kodlamayan kısımları (intronlar) çıkartılır, ayrıca mRNA'nın iki ucuna, onu nükleazlardan koruyucu eklemeler yapılır. Bunun ardından mRNA çekirdekten sitoplazmaya taşınır, orada ribozomlara bağlanır ve tRNA'nın yardımıyla çevirisi (translasyonu) yapılır. Prokaryotlarda, çekirdek olmadığından, RNA'nın transkripsiyonu sürerken ribozomlar tarafından çevirisi başlar. Bir süre sonra mesajcı RNA ribonükleazlar tarafından parçalanır.[22]
Taşıyıcı RNA (tRNA) yaklaşık 80 nükleotit uzunluğunda bir RNA zinciri olup, ribozomun protein sentez konumunda büyümekte olan polipeptide spesifik aminoasitler taşır. Yapısında, mRNA'daki kodonları tanımak için onlarla hidrojen bağı kuran bir antikodon bölgesi ve amino asidin ona bağlanması için gerekli bölgeler vardır.[23]
Ribozomal RNA (rRNA) ribozomların katalitik kısmıdır. Ökaryotik ribozomlar dört RNA içerirler: 18S, 5.8S, 28S and 5S rRNA. Bu rRNA'lardan üçü çekirdekçikte sentezlenir. Sitoplazmada ribozomal RNA ve proteinler bir araya gelip ribozomu oluştururlar. Ribozom mRNA'ya bağlanır ve protein sentezini gerçekleştirir. Bir mRNA'ya aynı andan birkaç yüz ribozom bağlanabilir.[22] Tipik bir ökaryotik hücre sitoplazmasındaki RNA konsantrasyonu 10 mg/ml'dir, bunun %80 rRNA'dan oluşur.[24]
Gen düzenlemesinde
Bazı RNA tipleri genin belli bir kısmının dizisine tümleyici olarak gen ifadesinin aşağı ayarlayabilirler. Ökaryotlarda bulunan mikro RNA'lar (miRNA; 21-22 nt) RNA enterferans yoluyla etki eder. RNA enterferansında miRNA ve enzimlerden oluşan bir kompleks, miRNA'nın tümleyici olduğu bir mRNA'yı parçalayabilir, veya mRNA'nın translasyonunu bloke edebilirler, veya promotörün metilasyonuna neden olarak genelde geni aşağı ayarlarlar.[25] Bazı miRNA'lar ise genleri yukarı ayarlarlar (RNA aktivasyonu).[26] Küçük enterferansçı RNA (İng. small ınterfering RNA, siRNA)'lar 20-25 nt uzunlukta olurlar, genelde viral RNA'nın parçalanmasından meydana gelmelerine karşın, bu RNA tiplerinin endojen kaynakları da mevcuttur.[27] siRNA'lar, miRNA'ya benzer şekilde )RNA aktivasyonu da dahil olmak üzere) RNA enterferansı aracılığyla etki ederler.[28] Hayvanlarda bulunan Piwi etkileşimli RNA'lar (İngilizce Piwi-interacting RNAs, piRNA; 29-30 nt) eşey hücrelerinde etkindirler, transpozonlara karşı savunmaya yaradıkları ve gametogenezde rol oynadıkları düşünülmektedir.[29][30] Dişi hayvanlarda görülen X kromozom inaktivasyonu, X kromozomlarından birini kaplayarak onu inaktive eden Xist adlı bir RNA tarafından meydana gelir.[31] Ters anlamlı RNA bakterilerde yaygındır; çoğu genleri aşağı ayarlar ama bazıları da transkripsiyon aktivatörüdür.[32] Bir mRNA'nın kendisi de 5 üssü çevrilmeyen bölgesinde veya 3 üssü çevrilmeyen bölgesinde riboanahtar gibi düzenleyici elemanlar içerebilir. Bu beri-düzenleyici unsurlar (İng. cis-regulatory element) mRNA'nın etkinliğini düzenlerler.[33]

RNA işlenmesinde
Çoğu RNA başka RNA'ların modifikasyonunda rol oynar. Örneğin uçbirleştirmede, pre-mRNA'daki intronların çıkartılmasını sağlayan splisozom, küçük nükleer RNA (snRNA)'lar içerir.[19]
RNA'yı oluşturan nükleotitlerler A, C, G ve U'dan farklı bazlara değişime uğrayabilir. Ökaryotlarda RNA nükleotitlerinin modifikasyonu genelde çekirdekçik ve Cajal cisimlerinde bulunan küçük nükleolar RNA (small nucleolar RNA, snoRNA; 60-300 nt) tarafından yönlendirilir.[23] SnoRNA'lar enzimlerle birleşip onları RNA üzerindeki belli bir noktaya yönlendiriler, bunu sağlamak için RNA ile baz eşleşmesi yaparlar. Bu enzimler sonra o noktadaki nükleotit modifikasyonunu gerçekleştirler. rRNA ve tRNA bu şekilde büyük oranda değişime uğrarlar, ama snRNA ve mRNA'ların da bu yolla modifiye oldukları görülmüştür,[34][35]
RNA tipleri
| Tip | Kısaltma | İşlev | Dağılım | Ref. |
|---|---|---|---|---|
| Mesajcı RNA | mRNA | Protein kodlaması. | Tüm canlılar | |
| Ribozomal RNA | rRNA | Protein sentezi | Tüm canlılar | |
| Taşıyıcı RNA | tRNA | Protein sentezi | Tüm canlılar | |
| Taşıyıcı-mesajcı RNA | tmRNA | Takılıp kalmış ribozomların kurtarılması | Bakteriler | [36] |
| Ters anlamlı RNA (İng. antisense RNA) | aRNA | Gen düzenlemesi | Tüm canlılar | [37] |
| Küçük enterferanscı RNA (İng. Small interfering RNA) | siRNA | Gen düzenlemesi | Çoğu ökaryot | [38] |
| Mikro RNA | miRNA | Gen düzenlemesi | Çoğu ökaryot | [39] |
| trans-etken siRNA (İng. trans-acting siRNA) | tasiRNA | Gen düzenlemesi | Bitkiler (Arabidopsis thaliana) | [40] |
| Piwi-etkileşimli RNA (İng. Piwi-interacting RNA) | piRNA | Gen düzenlemesi | Hayvanlar | [29] |
| küçük nükleer RNA (İng. Small nuclear RNA) | snRNA | Çeşitli | Ökaryotlar ve arkeler | [41] |
| Küçük nükleolar RNA (İng. Small nucleolar RNA) | snoRNA | RNA'nın çekirdekte modifikasyonu | Ökaryotlar ve arkeler | [42] |
| Yönledirici RNA (İng. Guide RNA) | gRNA | mRNA modifikasyonu | Kinetoplastid mitokondrileri | [43] |
| Ribonükleaz P | RNaz P | tRNA erginleşmesi | Tüm canlılar | [44] |
| Ribonükleaz MRP | RNaz MRP | rRNA erginleşmesi, DNA replikasyonu | Ökaryotlar | [45] |
| Y RNA | RNA işlenmesi, DNA replikasyonu | Hayvanlar | [46] | |
| Telomeraz RNA | Telomer sentezi | Çoğu ökaryot | [47] | |
| Sinyal tanıma taneciği RNA'sı (Signal recognition particle RNA) | SRP RNA | Protein ihracı | Tüm canlılar | [48] |
| Retrotranspozon | kendini çoğaltmak | Ökaryotlar ve bazı bakteriler | [49] | |
| Viroid | kendini çoğaltmak | Enfekte bitkiler | [50] | |
| Not: Bu liste Szymanski et al. 2003.[51] makalesi esas alınmıştır. | ||||
Yukarıda belirtilenlere ilaveten, pek çok virüs genomu RNA'dan oluşur. Bunlar çift iplikli RNA virüsleri, pozitif anlamlı tek zincirli RNA virüsler, negatif anlamlı tek zincirli RNA virüsleri ve çoğu satelit virüslerdir.
Keşif
Nükleik asitler 1868'de Friedrich Miescher tarafından keşfedilmiş, hücre çekirdeğinde (nucleusta) yer aldığı için Miescher bu maddeye 'nüklein' adını vermişti.[52] Daha sonradan nükleik asitlerin çekirdeksiz olan prokaryotlarda da olduğu bulunmuştu. RNA'nın protein sentezinde rol oynadığı 1939'ten itibaren, Torbjörn Caspersson, Jean Brachet ve Jack Schultz'un deney sonuçlarından dolayı, tahmin edilmekteydi.[53] Gerard Marbaix ilk mesajcı RNA'yı (tavşan hemoglobinine ait olan) saflaştırmış, ve onu yumurta hücrelerine enjekte edince bunun hemoglobin sentezini sağladığını göstermişti.[54] Severo Ochoa RNA'nın nasıl sentezlendiğini keşfettikten sonra 1950 Nobel Tıp Ödülünü kazandı.[55] Robert W. Holley bir maya RNA'sının ilk 77 nükleotidinin dizisini 1965'te çözmüş,[56] bundan dolayı 1968 Nobel Tıp ödülünü kazanmıştır. Carl Woese ve diğerleri 1967'de RNA'nın katalitik olduğunu buldular[57] en eski canlı tiplerinin bir "RNA Dünyası" içinde yaşadıklarını, RNA'yı hem genetik bilgi taşımak hem de biyokimyasal tepkimeleri katalizlemek için kullanmış olabileceğini öne sürdüler.[58] 1976'da Walter Fiers ve arkadaşları ilk defa bir RNA virüs genomunun (bakteriyofaj MS2'nin) tüm nükleotit dizisini belirlediler.[59]
1990 başlarında bitki hücrelerinin içine sokulan genlerin bunlara benzer endojen genleri susturduğu bulundu.[60] Yaklaşık aynı dönemde, 22 nt uzunlukta (günümüzde mikroRNA olarak adlandırılan) RNA'ların C. elegans solucanının gelişimine etki ettiği keşfedildi.[61]
Gen düzenleyici RNA'ların keşfi üzerine, onkogenleri ve viral genleri susturabilecek RNA'dan oluşmuş ilaçlar geliştirmeye yönelik çabalar başladı.[62]. 2006 itibariyle piyasada bu özellikli tek bir ilaç bulunmaktadır, bir sitomegalovirüs genini inhibe etmeye yarayan Vitravene (bir ters anlamlı RNA), ama RNA enterferans yoluyla genleri aşağı ayarlamak için siRNA kullanmaya yönelik ümit verici araştırmalar sürmektedir.[63]
Kaynakça
- 1 2 Lee JC, Gutell RR (2004). "Diversity of base-pair conformations and their occurrence in rRNA structure and RNA structural motifs". J. Mol. Biol. 344 (5): 1225–49. DOI:10.1016/j.jmb.2004.09.072. PMID 15561141.
- ↑ Barciszewski J, Frederic B, Clark C (1999). RNA biochemistry and biotechnology. Springer. s. 73–87. ISBN 0792358627.
- ↑ Salazar M, Fedoroff OY, Miller JM, Ribeiro NS, Reid BR (1992). "The DNA strand in DNAoRNA hybrid duplexes is neither B-form nor A-form in solution". Biochemistry 1993 (32): 4207–15. PMID 7682844.
- ↑ Hermann T, Patel DJ (2000). "RNA bulges as architectural and recognition motifs". Structure 8 (3): R47–R54. DOI:10.1016/S0969-2126(00)00110-6.
- ↑ Mikkola S, Nurmi K, Yousefi-Salakdeh E, Strömberg R, Lönnberg H (1999). "The mechanism of the metal ion promoted cleavage of RNA phosphodiester bonds involves a general acid catalysis by the metal aquo ion on the departure of the leaving group". Perkin transactions 2: 1619–26. DOI:10.1039/a903691a.
- ↑ Jankowski JAZ, Polak JM (1996). Clinical gene analysis and manipulation: tools, techniques and troubleshooting. Cambridge University Press. s. 14. ISBN 0521478960.
- ↑ Yu Q, Morrow CD (2001). "Identification of critical elements in the tRNA acceptor stem and TΨC loop necessary for human immunodeficiency virus type 1 infectivity". J Virol. 75 (10): 4902–6. DOI:10.1128/JVI.75.10.4902-4906.2001.
- ↑ Elliott MS, Trewyn RW (1983). "Inosine biosynthesis in transfer RNA by an enzymatic insertion of hypoxanthine". J. Biol. Chem. 259 (4): 2407–10. PMID 6365911.
- ↑ Söll D, RajBhandary U (1995). tRNA: Structure, biosynthesis, and function. ASM Press. s. 165. ISBN 155581073X.
- ↑ Kiss T (2001). "Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs". The EMBO Journal 20: 3617–22. DOI:10.1093/emboj/20.14.3617.
- ↑ King TH, Liu B, McCully RR, Fournier MJ (2002). "Ribosome structure and activity are altered in cells lacking snoRNPs that form pseudouridines in the peptidyl transferase center". Molecular Cell 11 (2): 425–35. DOI:10.1016/S1097-2765(03)00040-6.
- ↑ Mathews DH, Disney MD,Childs JL, Schroeder SJ, Zuker M, Turner DH (2004). "Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure". Proc. Natl. Acad. Sci. USA 101 (19): 7287–92. DOI:10.1073/pnas.0401799101.
- ↑ Higgs PG (2000). "RNA secondary structure: physical and computational aspects". Quarterly Reviews of Biophysics 33: 199–253. DOI:10.1017/S0033583500003620.
- 1 2 Nissen P, Hansen J, Ban N, Moore PB, Steitz TA (2000). "The structural basis of ribosome activity in peptide bond synthesis". Science 289 (5481): 920–30. DOI:10.1126/science.289.5481.920.
- ↑ Nudler E, Gottesman ME (2002). "Transcription termination and anti-termination in E. coli". Genes to Cells 7: 755–68. DOI:10.1046/j.1365-2443.2002.00563.x.
- ↑ Jeffrey L Hansen, Alexander M Long, Steve C Schultz (1997). "Structure of the RNA-dependent RNA polymerase of poliovirus". Structure 5 (8): 1109-22. DOI:10.1016/S0969-2126(97)00261-X.
- ↑ Ahlquist P (2002). "RNA-Dependent RNA Polymerases, Viruses, and RNA Silencing". Science 296 (5571): 1270–73. DOI:10.1126/science.1069132.
- ↑ Nelson C. Lau, Lee P. Lim, Earl G. Weinstein, David P. Bartel (2001). "An Abundant Class of Tiny RNAs with Probable Regulatory Roles in Caenorhabditis elegans". Science 294 (5543): 858-862. DOI:10.1126/science.1065062. PMID 11679671.
- 1 2 Berg JM, Tymoczko JL, Stryer L (2002). Biochemistry (5th edition bas.). WH Freeman and Company. s. 118–19, 781–808. ISBN 0-7167-4684-0.
- ↑ Rossi JJ (2004). "Ribozyme diagnostics comes of age". Chemistry & Biology 11 (7): 894–95. DOI:10.1016/j.chembiol.2004.07.002.
- ↑ Blevins T et al (2006). "Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing". Nucleic Acids Res 34 (21): 6233–46. PMID 17090584.
- 1 2 Cooper GC, Hausman RE (2004). The Cell: A Molecular Approach (3rd edition bas.). Sinauer. s. 261–76, 297, 339–44. ISBN 0-87893-214-3.
- 1 2 Wirta W (2006). Mining the transcriptome – methods and applications. ISBN 91-7178-436-5. http://www.diva-portal.org/diva/getDocument?urn_nbn_se_kth_diva-4115-3__fulltext.pdf.
- ↑ Kampers T, Friedhoff P, Biernat J, Mandelkow E-M, Mandelkow E (1996). "RNA stimulates aggregation of microtubule-associated protein tau into Alzheimer-like paired helical filaments". FEBS Letters 399: 98–100, 344–49. PMID 8985176.
- ↑ Matzke MA, Matzke AJM (2004). "Planting the seeds of a new paradigm". PLoS Biology 2 (5): e133. DOI:10.1371/journal.pbio.0020133.
- ↑ Check E (2007). "RNA interference: hitting the on switch". Nature 448 (7156): 855–58. DOI:10.1038/448855a.
- ↑ Vazquez F, Vaucheret H, Rajagopalan R, Lepers C, Gasciolli V, Mallory AC, Hilbert J, Bartel DP, Crété P (2004). "Endogenous trans-acting siRNAs regulate the accumulation of Arabidopsis mRNAs". Molecular Cell 16 (1): 69–79. DOI:10.1016/j.molcel.2004.09.028.
- ↑ Doran G (2007). "RNAi – Is one suffix sufficient?". Journal of RNAi and Gene Silencing 3 (1): 217–19. http://libpubmedia.co.uk/RNAiJ-Issues/Issue-5/Doran.htm.
- 1 2 Horwich MD, Li C Matranga C, Vagin V, Farley G, Wang P, Zamore PD (2007). "The Drosophila RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC". Current Biology 17: 1265–72. DOI:10.1016/j.cub.2007.06.030.
- ↑ Girard A, Sachidanandam R, Hannon GJ, Carmell MA (2006). "A germline-specific class of small RNAs binds mammalian Piwi proteins". Nature 442: 199–202. DOI:10.1038/nature04917.
- ↑ Heard E, Mongelard F, Arnaud D, Chureau C, Vourc'h C, Avner P (1999). "Human XIST yeast artificial chromosome transgenes show partial X inactivation center function in mouse embryonic stem cells". Proc. Natl. Acad. Sci. USA 96 (12): 6841–46. PMID 10359800.
- ↑ Wagner EG, Altuvia S, Romby P (2002). "Antisense RNAs in bacteria and their genetic elements". Adv Genet. 46: 361–98. PMID 11931231.
- ↑ Batey RT (2006). "Structures of regulatory elements in mRNAs". Curr. Opin. Struct. Biol. 16 (3): 299–306. DOI:10.1016/j.sbi.2006.05.001. PMID 16707260.
- ↑ Covello PS, Gray MW (1989). "RNA editing in plant mitochondria". Nature 341: 662–66. DOI:10.1038/341662a0.
- ↑ Omer AD, Ziesche S, Decatur WA, Fournier MJ, Dennis PP (2003). "RNA-modifying machines in archaea". Molecular Microbiology 48 (3): 617–29. DOI:10.1046/j.1365-2958.2003.03483.x.
- ↑ Gillet R, Felden B (2001). "Emerging views on tmRNA-mediated protein tagging and ribosome rescue". Molecular Microbiology 42 (4): 879–85. DOI:10.1046/j.1365-2958.2001.02701.x.
- ↑ Brantl S (2002). "Antisense-RNA regulation and RNA interference". Biochimica et Biophysica Acta 1575 (1–3): 15–25. PMID 12020814.
- ↑ Ahmad K, Henikoff S (2002). "Epigenetic consequences of nucleosome dynamics". Cell 111 (3): 281–84. DOI:10.1016/S0092-8674(02)01081-4.
- ↑ Lin S-L, Miller JD, Ying S-Y (2006). "Intronic microRNA (miRNA)". Journal of Biomedicine and Biotechnology: 1–13. PMID 17057362.
- ↑ Vazquez F, Vaucheret H (2004). "Endogenous trans-acting siRNAs regulate the accumulation of Arabidopsis mRNAs". Mol. Cell (16): 1–13. PMID 17057362.
- ↑ Thore S, Mayer C, Sauter C, Weeks S, Suck D (2003). "Crystal Structures of the Pyrococcus abyssi Sm Core and Its Complex with RNA". J. Biol. Chem. 278 (2): 1239–47. DOI:10.1074/jbc.M207685200.
- ↑ Kiss T (2001). "Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs". The EMBO Journal 20: 3617–22. DOI:10.1093/emboj/20.14.3617.
- ↑ Alfonzo JD, Thiemann O, Simpson L (1997). "The mechanism of U insertion/deletion RNA editing in kinetoplastid mitochondria". Nucleic Acids Research 25 (19): 3751–59. PMID 9380494.
- ↑ Pannucci JA, Haas ES, Hall TA, Harris JK, Brown JW (1999). "RNase P RNAs from some Archaea are catalytically active". Proc Natl Acad Sci USA 96 (14): 7803–08. PMID 10393902.
- ↑ Woodhams MD, Stadler PF, Penny D, Collins LJ (2007). "RNase MRP and the RNA processing cascade in the eukaryotic ancestor". BMC Evolutionary Biology 7: S13. DOI:10.1186/1471-2148-7-S1-S13.
- ↑ Perreault J, Perreault J-P, Boire G (2007). "Ro-associated Y RNAs in metazoans: evolution and diversification". Molecular Biology and Evolution 24 (8): 1678–89. DOI:10.1093/molbev/msm084.
- ↑ Lustig AJ (1999). "Crisis intervention: The role of telomerase". Proc Natl Acad Sci USA 96 (7): 3339–41. PMID 10097039.
- ↑ Gribaldo1 S, Brochier-Armanet C (2006). "The origin and evolution of Archaea: a state of the art". Philos Trans R Soc Lond B Biol Sci. 361 (1470): 1007–22. PMID 16754611.
- ↑ Boeke JD (2003). "The unusual phylogenetic distribution of retrotransposons: a hypothesis". Genome Research 13: 1975–83. PMID 12952870.
- ↑ Flores R, Hernández C, Martínez de Alba AE, Daròs JA, Di Serio F (2005). "Viroids and viroid-host interactions". Annual Review of Phytopathology 43: 117–39. PMID 16078879.
- ↑ Szymanski J, Barciszewska MZ, Zywicki M, Barciszewski J (2003). "Noncoding RNA transcripts". J. Appl. Genet. 44 (1): 9. PMID 12590177.
- ↑ Dahm R (2005). "Friedrich Miescher and the discovery of DNA". Developmental Biology 278 (2): 274–88. PMID 15680349.
- ↑ Nierhaus KH, Wilson DN (2004). Protein Synthesis and Ribosome Structure. Wiley-VCH. s. 3. ISBN 3527306382.
- ↑ Carlier M (June 2003). "L'ADN, cette "simple" molécule". Esprit libre. 3 Mart 2016 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20160303185328/http://www.ulb.ac.be/espritlibre/html/el062003/32.html.
- ↑ Ochoa S (1959). "Enzymatic synthesis of ribonucleic acid". Nobel Lecture. 4 Mart 2016 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20160304083303/http://www.nobelprize.org/nobel_prizes/medicine/laureates/1959/ochoa-lecture.pdf.
- ↑ Holley RW et al (1965). "Structure of a ribonucleic acid". Science 147 (1664): 1462–65. DOI:10.1126/science.147.3664.1462.
- ↑ Siebert S (2006). "Common sequence structure properties and stable regions in RNA secondary structures". Dissertation, Albert-Ludwigs-Universität, Freiburg im Breisgau. ss. 1. 9 Mart 2012 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20120309212648/http://deposit.ddb.de/cgi-bin/dokserv?idn=982323891&dok_var=d1&dok_ext=pdf&filename=982323891.pdf.
- ↑ Szathmáry E (1999). "The origin of the genetic code: amino acids as cofactors in an RNA world". Trends Genet. 15 (6): 223–9. DOI:10.1016/S0168-9525(99)01730-8.
- ↑ Fiers W et al (1976). "Complete nucleotide-sequence of bacteriophage MS2-RNA: primary and secondary structure of replicase gene". Nature 260: 500–7. PMID 1264203.
- ↑ Napoli C, Lemieux C, Jorgensen R (1990). "Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans.". Plant Cell 2 (4): 279–89. PMID 12354959.
- ↑ Ruvkun G (2001). "Glimpses of a tiny RNA world". Science 294 (5543): 797–99. DOI:10.1126/science.1066315.
- ↑ Trent RJ (2005). Molecular Medicine (3rd edition bas.). Elsevier. s. 160–63. ISBN 0-12-699057-3.
- ↑ Fichou Y, Férec C (2006). "The potential of oligonucleotides for therapeutic applications". Trends in Biotechnology 24 (12): 563–70. DOI:10.1016/j.tibtech.2006.10.003.
Dış bağlantılar
- RNA World RNA Dünyası sitesi (İngilizce)
- Nucleic Acid Database (Nükleik asit veritabanı) DNA, RNA ve komplekslerinin resimleri. (İngilizce)
- RNAJunction Database RNA eklemleri ve "öpüşen halka" yapılarının modelleri. (İngilizce)
| ||||||||||||||||||||